# meu computador (mudar de acordo)
setwd("D:/Desktop/UFSC/aulas/classes/RGV410046/data")4. Seleção e filtragem
Diretório
Pacotes e dados
library(rio)
library(tidyverse)
library(metan)Introdução
Após seus dados estarem carregados no ambiente R, eles provavelmente necessitarão de alguma manimulação antes de serem utilizados em uma determinada análise. Esta manipulação pode envolver operações como exclusão de colunas, ordenamento de linhas com base em valores, criação de covariáveis (que serão resultado de operações com uma ou mais variáveis existentes), dentre muitas outras possibilidades. Felizmente, o pacote dplyr permite que esta manimulação seja relativamente fácil, lógica (do ponto de vista de digitação de códigos) e rápida, pois ele integra a linguagem C++ em suas funções.
O pacote dplyr é uma gramática de manipulação de dados. Nos rerferimos à gramática aqui porque ele fornece funções nomeadas como verbos simples, relacionados às tarefas mais comuns de manipulação de dados, para ajudá-lo a traduzir seus pensamentos em código. Este será o pacote utilizado para manipulação dos dados no decorrer deste material. De fato, a maioria dos dados em R podem ser manipulados utilizando os seguintes “verbos”.
select()erename()para selecionar variáveis com base em seus nomes.mutate()etransmute()para adicionar novas variáveis que são funções de variáveis existentes.arrange()para reordenar as linhas.filter()para selecionar linhas com base em seus valores.slice_*()para selecionar linhas com base em sua posição.summarise()para resumir vários valores para um único valor.group_by()para agrupar dados com base em níveis de variáveis categóricas.across()para aplicar a mesma transformação a várias colunas.
Anteriomente mencionamos que a manipulação dos dados com o pacote dplyr é lógica do ponto de vista da implementação do código. Isto só é possivel devido a utilização do operador %>% (forward-pipe operator), importado do pacote magrittr (ou com o pipe |> disponível do R vesão 4.1 ou superior). Basicamente, este operador capta o argumento resultante de uma função à sua esquerda e passa como input à função à sua direita. Considere as seguintes (e simples) operações. Crie um data frame com 100 linhas com as variáveis x e y contendo valores aleatórios. Adicione uma terceira variáveis (z) que será uma função da multiplicação de x e y, selecione apenas os valores de z menores que 10 e extraia a raiz quadrada destes valores. Finalmente, compute a média e armazene no object mean_sqrt.
- Criando o conjunto de dados
set.seed(1)
data <-
tibble(x = runif(100, 0, 10),
y = runif(100, 0, 10))- Utilizando as funções bases do R (código massivo)
data$z <- data$x * data$y
df <- subset(data, z < 10)
df <- df[, 3]
sqr_dat <- sqrt(df$z)
mean_sqrt <- mean(sqr_dat)
mean_sqrt[1] 1.977507- Utilizando as funções bases do R (código mais limpo, envelopando funções)
data$z <- data$x * data$y
mean_sqrt <- mean(sqrt(subset(data, z < 10)$z))
mean_sqrt[1] 1.977507- Utilizando o operdor
|>
mean_sqrt <-
data |>
mutate(z = x * y) |>
filter(z < 10) |>
pull(z) |>
sqrt() |>
mean()
mean_sqrt[1] 1.977507Trabalhando com colunas
Selecionar colunas
A função select() do pacote dplyr pode ser usada para selecionar colunas de um conjunto de dados com base em seu nome (por exemplo, a:f seleciona todas as colunas de a à esquerda a f à direita). Você também pode usar funções de predicado como is.numeric para selecionar variáveis com base em suas propriedades. As seleções do Tidyverse implementam um dialeto R onde os operadores facilitam a seleção de variáveis:
:para selecionar um intervalo de variáveis consecutivas.!para tomar o complemento de um conjunto de variáveis.&e|para selecionar a interseção ou a união de dois conjuntos de variáveis.c()para combinar seleções.
Com base em seus nomes
maize <-
import("examples_data.xlsx",
sheet = "maize",
setclass = "tbl")
# para evitar uma saída longa
df <- maize |> slice(1:5)
# lista de nomes
df |> select(AMB, HIB, REP)# A tibble: 5 × 3
AMB HIB REP
<chr> <chr> <chr>
1 A1 H1 I
2 A1 H1 I
3 A1 H1 I
4 A1 H1 I
5 A1 H1 I # sequência de nomes
df |> select(AMB:REP)# A tibble: 5 × 3
AMB HIB REP
<chr> <chr> <chr>
1 A1 H1 I
2 A1 H1 I
3 A1 H1 I
4 A1 H1 I
5 A1 H1 I # vector de posições
df |> select(1:3)# A tibble: 5 × 3
AMB HIB REP
<chr> <chr> <chr>
1 A1 H1 I
2 A1 H1 I
3 A1 H1 I
4 A1 H1 I
5 A1 H1 I # negar a seleção
df |> select(!c(AMB:REP))# A tibble: 5 × 7
APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2.45 2.39 16.9 52.1 228. 375. NA
2 2.5 1.43 14.4 50.7 187. 437. 427
3 2.69 1.52 16.5 54.7 230. 464. 497
4 2.8 1.64 16.8 52.0 213. 408. 523
5 2.62 1.55 15.9 51.6 224. 406. 551Com base na classe
A função where() aplica uma função a todas as variáveis e seleciona aquelas para as quais a função retorna TRUE. Assim, podemos selecionar facilmente colunas com base em sua classe
# seleciona variáveis numéricas
df |> select(where(is.numeric))# A tibble: 5 × 7
APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2.45 2.39 16.9 52.1 228. 375. NA
2 2.5 1.43 14.4 50.7 187. 437. 427
3 2.69 1.52 16.5 54.7 230. 464. 497
4 2.8 1.64 16.8 52.0 213. 408. 523
5 2.62 1.55 15.9 51.6 224. 406. 551# seleciona variáveis não numéricas
df |> select(!where(is.numeric))# A tibble: 5 × 3
AMB HIB REP
<chr> <chr> <chr>
1 A1 H1 I
2 A1 H1 I
3 A1 H1 I
4 A1 H1 I
5 A1 H1 I Select helpers
Essas funções permitem selecionar variáveis com base em seus nomes.
starts_with(): começa com um prefixo
df |> select(starts_with("C"))# A tibble: 5 × 1
CESP
<dbl>
1 16.9
2 14.4
3 16.5
4 16.8
5 15.9ends_with(): termina com um prefixo
df |> select(ends_with("S"))# A tibble: 5 × 1
DIES
<dbl>
1 52.1
2 50.7
3 54.7
4 52.0
5 51.6# variáveis que começam com M e terminam com A
df |> select(starts_with("M") & ends_with("A"))# A tibble: 5 × 1
MGRA
<dbl>
1 228.
2 187.
3 230.
4 213.
5 224.# variáveis que começam com M ou terminam com A
df |> select(starts_with("M") | ends_with("A"))# A tibble: 5 × 3
MGRA MMG NGRA
<dbl> <dbl> <dbl>
1 228. 375. NA
2 187. 437. 427
3 230. 464. 497
4 213. 408. 523
5 224. 406. 551contains(): contém uma string literal
Se as variáveis no conjunto de dados tiverem um padrão com diferenças entre um grupo de variáveis, podemos usar o código a seguir para selecionar variáveis com um padrão.
df |> select(contains("PLANT"))# A tibble: 5 × 2
APLA_PLANT AIES_PLANT
<dbl> <dbl>
1 2.45 2.39
2 2.5 1.43
3 2.69 1.52
4 2.8 1.64
5 2.62 1.55matches(): corresponde a uma expressão regular
Seleções mais sofisticadas podem ser feitas usando matches(). Supondo que gostaríamos de selecionar as variáveis que começam com “A” e tem a segunda letra entre “A” e “M”, usaríamos algo como
df |> select(matches("^A[A-M]"))# A tibble: 5 × 2
AMB AIES_PLANT
<chr> <dbl>
1 A1 2.39
2 A1 1.43
3 A1 1.52
4 A1 1.64
5 A1 1.55one_of(): variáveis no vetor de caracteres.
vars <- c("TESTE", "CESP", "NGRA", "NAO_TEM")
df |> select(one_of(vars))Warning: Unknown columns: `TESTE`, `NAO_TEM`# A tibble: 5 × 2
CESP NGRA
<dbl> <dbl>
1 16.9 NA
2 14.4 427
3 16.5 497
4 16.8 523
5 15.9 551everything(): todas as variáveis.
df |> select(everything())# A tibble: 5 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. NA
2 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
3 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497
4 A1 H1 I 2.8 1.64 16.8 52.0 213. 408. 523
5 A1 H1 I 2.62 1.55 15.9 51.6 224. 406. 551Extrair colunas
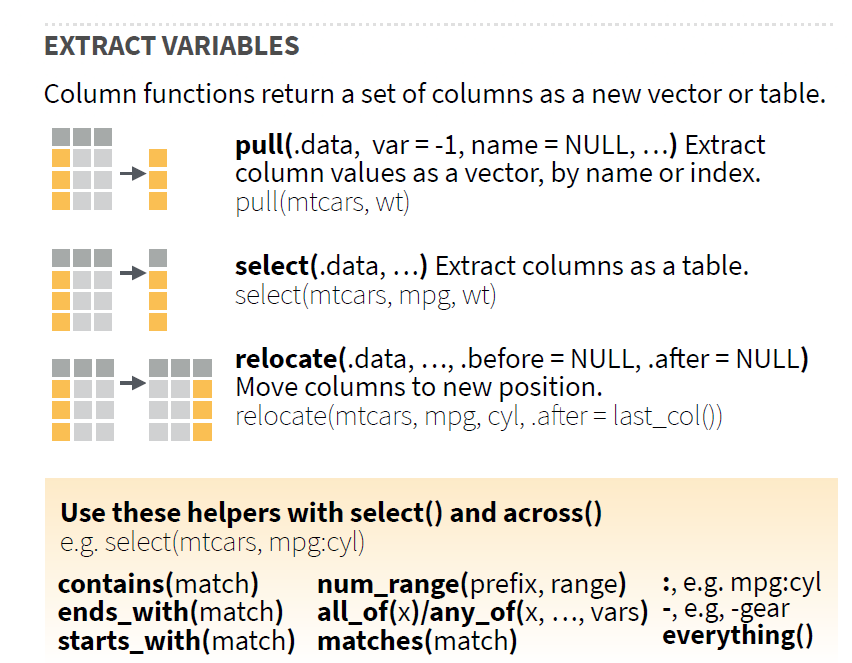
No R base, para extrair colunas de um data frame usamos $. A função pull() é semelhante a $, mas é mais fácil de ser utilizada com pipes. Para seleção, podemos especificar uma variável como:
- um nome de variável literal
- um inteiro positivo, dando a posição contando a partir da esquerda
- um inteiro negativo, dando a posição contando a partir da direita.
- O padrão retorna a última coluna (supondo que seja a coluna que você criou mais recentemente).
Note a diferença.
df$MGRA[1] 228.3716 186.6627 230.3904 213.4960 223.6949# padrão é a última coluna
df |> pull()[1] NA 427 497 523 551# selecionar variável com base no nome
df |> pull(MGRA)[1] 228.3716 186.6627 230.3904 213.4960 223.6949# selecionar variável com base na sua posição
df |> pull(5)[1] 2.39 1.43 1.52 1.64 1.55Realocar colunas (Avançado)
Para reordenar colunas em um data frame, podemos utilizar a função relocate() do pacote dplyr. Ela altera as posições das colunas, usando a mesma sintaxe que select() para facilitar a movimentação de blocos de colunas de uma só vez.
relocate(.data, ..., .before = NULL, .after = NULL)Nesta função, as variáveis em … são movidas para antes de .before ou depois de .after.
df |> relocate(NGRA, .before = APLA_PLANT)# A tibble: 5 × 10
AMB HIB REP NGRA APLA_PLANT AIES_PLANT CESP DIES MGRA MMG
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I NA 2.45 2.39 16.9 52.1 228. 375.
2 A1 H1 I 427 2.5 1.43 14.4 50.7 187. 437.
3 A1 H1 I 497 2.69 1.52 16.5 54.7 230. 464.
4 A1 H1 I 523 2.8 1.64 16.8 52.0 213. 408.
5 A1 H1 I 551 2.62 1.55 15.9 51.6 224. 406.df |> relocate(contains("_PLANT"), .after = last_col())# A tibble: 5 × 10
AMB HIB REP CESP DIES MGRA MMG NGRA APLA_PLANT AIES_PLANT
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 16.9 52.1 228. 375. NA 2.45 2.39
2 A1 H1 I 14.4 50.7 187. 437. 427 2.5 1.43
3 A1 H1 I 16.5 54.7 230. 464. 497 2.69 1.52
4 A1 H1 I 16.8 52.0 213. 408. 523 2.8 1.64
5 A1 H1 I 15.9 51.6 224. 406. 551 2.62 1.55df |> relocate(where(is.numeric), .before = where(is.character))# A tibble: 5 × 10
APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA AMB HIB REP
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <chr> <chr>
1 2.45 2.39 16.9 52.1 228. 375. NA A1 H1 I
2 2.5 1.43 14.4 50.7 187. 437. 427 A1 H1 I
3 2.69 1.52 16.5 54.7 230. 464. 497 A1 H1 I
4 2.8 1.64 16.8 52.0 213. 408. 523 A1 H1 I
5 2.62 1.55 15.9 51.6 224. 406. 551 A1 H1 I Trabalhado com linhas
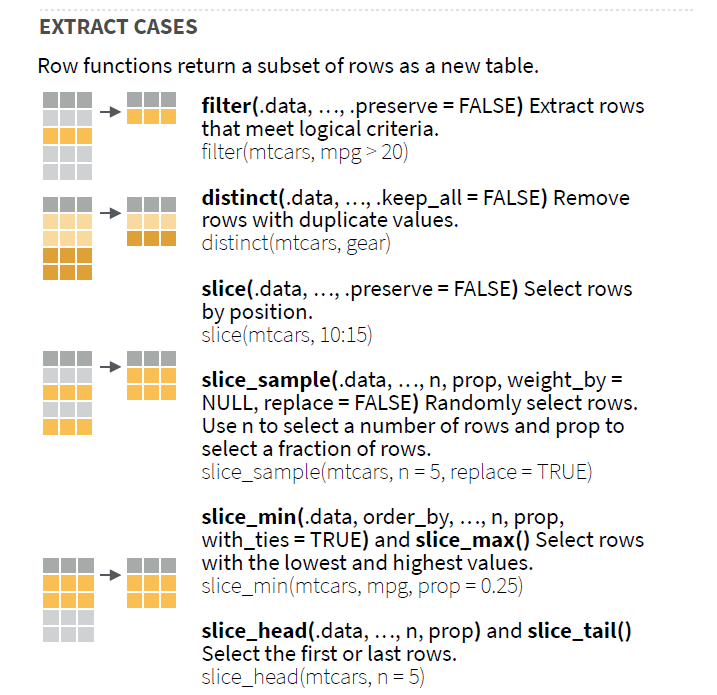
Selecionar linhas com base em seus valores
Utilizando a função filter() é possivel filtrar as linhas de um conjunto de dados com base no valor de suas variáveis. No primeiro exemplo, selecionaremos as linhas onde o valor da variável MGRA é maior que 280.
maize %>%
filter(MGRA > 280)# A tibble: 4 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H6 I 2.92 1.64 18 56.0 289. 393. 734
2 A1 H10 I 2.92 1.61 20.3 55.4 283. 441. 641
3 A1 H13 II 2.47 1.28 15.3 53.0 291. 417. 698
4 A4 H10 I 2.65 1.47 14 50.3 287. 275. 493No segundo exemplo, selecionaremos apenas as linhas onde a MGRA é maior que 220 OU a APLA é menor que 1.3 OU o NGRA é maior que 820.
maize %>%
filter(MGRA > 280 | APLA_PLANT < 1.3 | NGRA > 820)# A tibble: 13 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H6 I 2.92 1.64 18 56.0 289. 393. 734
2 A1 H10 I 2.92 1.61 20.3 55.4 283. 441. 641
3 A1 H13 II 2.47 1.28 15.3 53.0 291. 417. 698
4 A2 H8 II 1.03 0.69 10.8 44.8 94.8 277. 342
5 A2 H10 III 1.09 0.92 15 47.6 166. 299. 555
6 A2 H13 I 0 1.26 15.1 51.4 173. 375. 462
7 A3 H2 III 0 1.25 17.8 51.6 196. 348. 562
8 A3 H5 II 0 0.95 14.4 49.7 135. 213. 635
9 A3 H10 I 1.04 0.71 14.8 45.5 112. 265. 423
10 A3 H11 I 1 0.65 14.5 43.6 120. 210. 571
11 A4 H8 I 2.65 1.67 18 50 277. 251. 903
12 A4 H8 I 2.95 1.7 18.6 52.9 249. 302. 824
13 A4 H10 I 2.65 1.47 14 50.3 287. 275. 493No último exemplo, selecionaremos apenas as linhas onde MGRA é maior que é maior que 220 E a APLA é menor que 2.
maize %>%
filter(MGRA > 220 & APLA_PLANT < 2)# A tibble: 1 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H6 II 1.97 1.63 17.1 54.7 230. 375. 614Isto é aproximadamente equivalente ao seguinte código R base.
maize[maize$MGRA > 220 & maize$APLA_PLANT < 2, ]Você também pode usar filter() para remover grupos inteiros. Por exemplo, o código a seguir elimina todas as linhas que contém o híbrido “H1”.
maize |> filter(HIB != "H1")# A tibble: 720 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H2 I 2.45 1.24 17.7 54.7 255. 422. 604
2 A1 H2 I 2.41 1.32 15 50.5 198. 369. 537
3 A1 H2 I 2.62 1.16 10 47.5 94.5 380. 249
4 A1 H2 I NA 1.18 16.1 52.3 213. 331. 643
5 A1 H2 I 2.65 1.21 16.6 53.2 228. 409. 558
6 A1 H2 II 2.95 1.55 14.5 49.2 178. 258. 689
7 A1 H2 II 2.95 1.39 15.5 54.7 233. 284. 818
8 A1 H2 II 2.92 1.4 14 46.7 174. 288. 602
9 A1 H2 II 2.86 1.34 16.1 55.3 273. 356. 767
10 A1 H2 II 2.84 1.35 15.5 51.6 204. 335. 607
# ℹ 710 more rows# seleciona somente os híbridos H1 e H2
maize |> filter(HIB %in% c("H1", "H2"))# A tibble: 120 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. NA
2 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
3 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497
4 A1 H1 I 2.8 1.64 16.8 52.0 213. 408. 523
5 A1 H1 I 2.62 1.55 15.9 51.6 224. 406. 551
6 A1 H1 II 2.12 1.8 15 51.4 203. 383. 529
7 A1 H1 II 3.15 1.78 10.9 NA 75.2 256. 294
8 A1 H1 II 2.97 1.84 15 53.4 204. 387. 528
9 A1 H1 II 3.1 1.78 13.6 50.8 187. 348. 538
10 A1 H1 II 3.02 1.6 16.3 53.9 250. 430. 582
# ℹ 110 more rowsSelecionar linhas com base em sua posição
A função slice() permite indexar linhas por seus locais (inteiros). Ele permite selecionar, remover e duplicar linhas. Ele é acompanhado por vários auxiliares para casos de uso comuns:
slice_head()eslice_tail()selecionam a primeira ou a última linha.slice_sample()seleciona linhas aleatoriamente.slice_min()eslice_max()selecionam linhas com valores mais altos ou mais baixos de uma variável.
# selciona as primeiras três linhas
maize |> slice(1:3)# A tibble: 3 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. NA
2 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
3 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497# cinco linhas aleatórias
maize |> slice_sample(n = 5)# A tibble: 5 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H9 I NA 1.76 11.9 51.0 125. 423. 295
2 A1 H6 I 2.91 1.47 17.1 57.3 280. 420. 665
3 A2 H2 II 3.06 1.91 15 54.4 198. 501. 396
4 A4 H2 II 2.7 1.52 16.8 50.0 203. 363. 559
5 A1 H13 III 3.06 1.93 14.4 54.0 183. 382. 478# dois menores valores de MGRA
maize |> slice_min(n = 2, MGRA)# A tibble: 2 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H9 III 2.72 1.54 11 42.8 58.5 295. 198
2 A2 H8 I 1.92 0.63 12.1 39.7 59.5 243. 245# maior valor de NGRA
maize |> slice_max(n = 1, NGRA)# A tibble: 1 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A4 H8 I 2.65 1.67 18 50 277. 251. 903Ordenar linhas com base em seus valores
A função arrange() é utilizada para ordenar as linhas de um tibble (ou data.frames) com base em uma expressão envolvendo suas variáveis.
# ordena as linhas com base na variável CESP (crescente)
maize |> arrange(CESP)# A tibble: 780 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H9 I NA 1.51 0.800 54.0 224. 420. 533
2 A1 H8 I 2.6 1.4 5.9 52.2 225. 383. 588
3 A3 H8 III 1.86 0.96 7.5 40.8 73.0 293. 249
4 A1 H5 II 2.83 1.82 8.2 43.2 94.6 326. 290
5 A4 H3 II 2.37 1.12 8.2 47.4 96.0 256. 375
6 A2 H9 I 2.02 1.11 8.7 46.7 61.2 312. 196
7 A4 H3 II 2.22 1.02 8.9 41.6 77.8 218. 357
8 A3 H1 III 2.45 1.29 9 51.0 78.9 503. 157
9 A2 H3 I 2.82 1.87 9.3 47.2 105. 294. 358
10 A1 H2 III 2.86 1.52 9.4 51.2 91.0 225. 405
# ℹ 770 more rows# ordena as linhas com base na variável CESP (decrescente)
maize |> arrange(desc(CESP))# A tibble: 780 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A4 H7 III 2.6 1.37 20.4 52 253. 408. 620
2 A1 H10 I 2.92 1.61 20.3 55.4 283. 441. 641
3 A3 H1 I 2 1.05 19.9 53.3 253. 444. 570
4 A1 H10 III 2.29 1.15 19.8 55.9 276. 411. 671
5 A3 H2 II 2.08 0.93 19.6 52.9 220. 384. 574
6 A3 H9 III 2.07 1 19.6 48.2 190. 307. 617
7 A4 H8 III 2.6 1.5 19.5 56.1 264. 402. 657
8 A2 H6 III 3.18 1.62 19.2 53.0 270. 382. 708
9 A4 H7 III 2.76 1.54 19.2 54.1 254. 417. 610
10 A1 H9 I 2.69 1.8 19 52.4 226. 428. 529
# ℹ 770 more rowsAo combinar a função group_by() com arrange() é possível realizar o ordenamento para cada nível de um determinado fator. No exemplo abaixo, a variável APLA é ordenada de maneira crescente para cada híbrido.
maize %>%
group_by(HIB) %>%
arrange(MGRA, .by_group = TRUE)# A tibble: 780 × 10
# Groups: HIB [13]
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 II 3.15 1.78 10.9 NA 75.2 256. 294
2 A3 H1 III 2.45 1.29 9 51.0 78.9 503. 157
3 A3 H1 I 2.13 1.05 11.6 47.0 89.5 300. 298
4 A3 H1 II 2.18 1.04 13 46.6 103. 351. 293
5 A3 H1 II 2.08 0.94 12 47.6 103. 334. 309
6 A3 H1 I 2.07 1.05 13.2 47.9 110. 293. 377
7 A3 H1 II 1.93 0.93 13 50.0 120. 276. 433
8 A2 H1 III 3.11 1.9 13 50.8 131. 402. 325
9 A4 H1 I 2.3 1.25 13.1 50.0 140. 230. 609
10 A3 H1 II 2.39 1.21 14 50.2 144. 331. 435
# ℹ 770 more rowsOutras funções úteis (Avançado)
Algumas funções do pacote metan podem ser úteis para trabalhar com dados faltantes. Abaixo, alguns exemplos são mostrados.
# seleciona linhas com NA
maize |> select_rows_na()Warning: Rows(s) with NAs: 1, 7, 19, 124, 125, 257, 258, 259, 318, 319, 549,
550, 551, 552# A tibble: 14 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. NA
2 A1 H1 II 3.15 1.78 10.9 NA 75.2 256. 294
3 A1 H2 I NA 1.18 16.1 52.3 213. 331. 643
4 A1 H9 I NA 1.76 11.9 51.0 125. 423. 295
5 A1 H9 I NA 1.51 0.800 54.0 224. 420. 533
6 A2 H5 I NA 1.45 16.4 47.5 187. 306. 610
7 A2 H5 I NA 1.42 16.9 47.4 192. 382. 504
8 A2 H5 I NA 1.55 14 48.3 163. 355. 458
9 A2 H9 I NA 0.83 13.2 41.8 114. 218. 521
10 A2 H9 I NA 1.1 14.2 43.9 119. 186. 640
11 A3 H11 II 2.3 1 NA 45.1 99.8 303. 329
12 A3 H11 II 2.12 1.03 NA 47.8 104. 276. 375
13 A3 H11 III 2.4 1.3 NA 49.6 125. 277. 451
14 A3 H11 III 2.58 1.28 NA 46.8 177. 338. 523# remove linhas com NA
maize |> remove_rows_na()Warning: Row(s) 1, 7, 19, 124, 125, 257, 258, 259, 318, 319, 549, 550, 551, 552
with NA values deleted.# A tibble: 766 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
2 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497
3 A1 H1 I 2.8 1.64 16.8 52.0 213. 408. 523
4 A1 H1 I 2.62 1.55 15.9 51.6 224. 406. 551
5 A1 H1 II 2.12 1.8 15 51.4 203. 383. 529
6 A1 H1 II 2.97 1.84 15 53.4 204. 387. 528
7 A1 H1 II 3.1 1.78 13.6 50.8 187. 348. 538
8 A1 H1 II 3.02 1.6 16.3 53.9 250. 430. 582
9 A1 H1 III 2.69 1.52 15.6 49.5 195. 369. 529
10 A1 H1 III 2.6 1.68 14.3 48.9 172. 344. 500
# ℹ 756 more rows# substitu NA por 0
maize |> replace_na()# A tibble: 780 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. 0
2 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
3 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497
4 A1 H1 I 2.8 1.64 16.8 52.0 213. 408. 523
5 A1 H1 I 2.62 1.55 15.9 51.6 224. 406. 551
6 A1 H1 II 2.12 1.8 15 51.4 203. 383. 529
7 A1 H1 II 3.15 1.78 10.9 0 75.2 256. 294
8 A1 H1 II 2.97 1.84 15 53.4 204. 387. 528
9 A1 H1 II 3.1 1.78 13.6 50.8 187. 348. 538
10 A1 H1 II 3.02 1.6 16.3 53.9 250. 430. 582
# ℹ 770 more rows# substitui NA pela média da coluna (cuidado!!!)
maize |> replace_na(replacement = "colmean")# A tibble: 780 × 10
AMB HIB REP APLA_PLANT AIES_PLANT CESP DIES MGRA MMG NGRA
<chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 A1 H1 I 2.45 2.39 16.9 52.1 228. 375. 512.
2 A1 H1 I 2.5 1.43 14.4 50.7 187. 437. 427
3 A1 H1 I 2.69 1.52 16.5 54.7 230. 464. 497
4 A1 H1 I 2.8 1.64 16.8 52.0 213. 408. 523
5 A1 H1 I 2.62 1.55 15.9 51.6 224. 406. 551
6 A1 H1 II 2.12 1.8 15 51.4 203. 383. 529
7 A1 H1 II 3.15 1.78 10.9 49.5 75.2 256. 294
8 A1 H1 II 2.97 1.84 15 53.4 204. 387. 528
9 A1 H1 II 3.1 1.78 13.6 50.8 187. 348. 538
10 A1 H1 II 3.02 1.6 16.3 53.9 250. 430. 582
# ℹ 770 more rowsdplyr vs R base
A tabela a seguir mostra uma tradução condensada entre os verbos dplyr e seus equivalentes R base. As seções a seguir descrevem cada operação com mais detalhes. Você aprende mais sobre os verbos dplyr em sua documentação e em For more vignette(“one-table”).
| dplyr | base |
|---|---|
arrange(df, x) |
df[order(x), , drop = FALSE] |
distinct(df, x) |
df[!duplicated(x), , drop = FALSE], unique() |
filter(df, x) |
df[which(x), , drop = FALSE], subset() |
mutate(df, z = x + y) |
df$z <- df$x + df$y, transform() |
pull(df, 1) |
df[[1]] |
pull(df, x) |
df$x |
rename(df, y = x) |
names(df)[names(df) == "x"] <- "y" |
relocate(df, y) |
df[union("y", names(df))] |
select(df, x, y) |
df[c("x", "y")], subset() |
select(df, starts_with("x")) |
df[grepl(names(df), "^x")] |
summarise(df, mean(x)) |
mean(df$x), tapply(), aggregate(), by() |
slice(df, c(1, 2, 5)) |
df[c(1, 2, 5), , drop = FALSE] |
Desafio
- Crie uma variável chamada
MGRA_kgqual será o resultado em quilogramas da massa de grãos. - Selecione somente as colunas
HIB,AMB,REPeMGRA_Kg. - Selecione somente as cinco linhas com maior valor de
MGRA_Kg. - Compute os valores máximos, mínimos e a média destas cinco observações.