Capítulo 4 Loops
Reescrever um código muitas vezes por necessidade de repetir um determinado procedimento seria bastante trabalhoso, além de precisarmos de mais tempo para isso. Por isso, o R tem algumas funções que fazem essas repetições para nós. Isso é muito comum e pode ser facilmente implementado pela função for(), while() e repeat().
A função for() repete o código indicado dentro de {} n vezes, sendo n o comprimento da sequência dentro dos parênteses.
j <- 5
for (i in 1:j){
k <- i * 2
print(k)
}
# [1] 2
# [1] 4
# [1] 6
# [1] 8
# [1] 10A função while() (que significa enquanto) repete o código dentro de {} enquanto alguma condição for verdadeira.
i <- 1
while (i <= 5){
print(i * 2)
i <- i + 1
}
# [1] 2
# [1] 4
# [1] 6
# [1] 8
# [1] 10Note que os dois últimos exemplos apresentam o mesmo resultado: o R vai retornar uma sequência i sendo i = 1:5, onde cada número será o resultado da multiplicação \(i \times 2\). No caso while(), precisamos mudar o valor de i para que a sequência continue até que a condição (i <= 5) for verdadeira. Em adição, precisamos declarar a variável (i = 1) antes para que o R possa testar a condição expressa dentro dos parênteses. No caso do for(), a sequência progride sem precisarmos fazer isso manualmente.
No último exemplo, utilizando repeat(), o R repetirá o código dentro de {} sem condições. Com isso, precisamos utilizar a combinação das funções if() e break() para informar ao programa quando o código deve parar de ser repetido.
i <- 1
repeat {
print(i * 2)
i <- i + 1
if(i > 5){
break()
}
}
# [1] 2
# [1] 4
# [1] 6
# [1] 8
# [1] 10Os Loops são importantes em estudos que utilizam reamostragens para realizar análises estatísticas. Através de reamostragnes é possível construir intervalos de confiança e testar hipóteses não paramétricas. Como exemplo, vamos demonstrar o teorema central do limite20. Para isto, criamos uma função (teor_lim()) que tem 4 argumentos: n o tamanho da amostra a ser considerada, namostra, min, max são os parâmetros da distribuição uniforme (veja ?runif). Para confecção dos dendrogramas, os pacotes ggplot2 e cowplot serão utilizados. Veja a seção 1.6 para maiores informações sobre a confecção de gráficos com estes pacotes.
library(ggplot2)
library(cowplot)
teor_lim <- function(n, namostra, min, max){
set.seed(100)
media <- data.frame(matrix(ncol = 1, nrow = n))
names(media) = "value"
for(j in 1:n){
media[j, 1] = mean(runif(namostra, min, max))
}
return(media)
}
teorema_limite <- list(
n20 <- teor_lim(n = 20, namostra = 100, min = 0, max = 10),
n200 <- teor_lim(n = 200, namostra = 100, min = 0, max = 10),
n2000 <- teor_lim(n = 2000, namostra = 100, min = 0, max = 10),
n20000 <- teor_lim(n = 20000, namostra = 100, min = 0, max = 10)
)
p <- lapply(teorema_limite, function(d){
ggplot(data = d, aes(x = value))+
geom_histogram(bins = 50,
col = "black",
size = 0.3,
aes(fill = ..count..))+
theme(legend.position = "none")+
labs(x = "Média", y = "Contagem")
})
plot_grid(plotlist = p,
labels = names(teorema_limite),
vjust = 2.5,
hjust = c(-1.7, -1.5, -1, -1))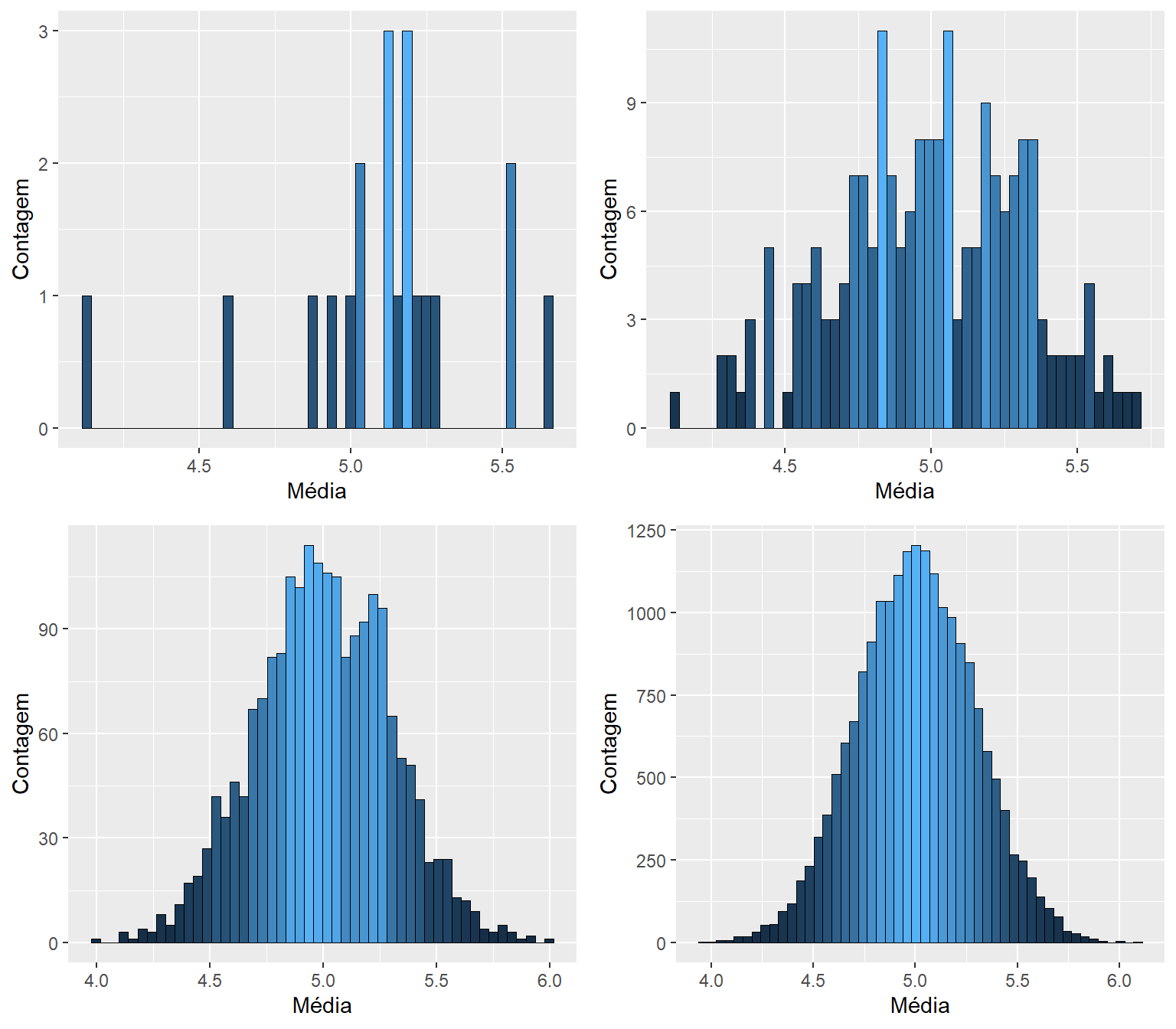
Figure 4.1: Demonstração do teorema central do limite